日本理化学研究所生命医科学研究中心基因组解析应用研究团队的小杉俊一研究员(现静冈县立综合医院研究支持中心遗传研究部研究员)、寺尾知可史组长(静冈县立综合医院临床研究部免疫研究部长)等人组成的共通研究团队发表研究成果称,开发出了一种在全基因组测序数据中高精度检测结构变异 (Structure Variantions,简称SV) 的新方法。相关成果已刊登在《Cell Genomics》上。
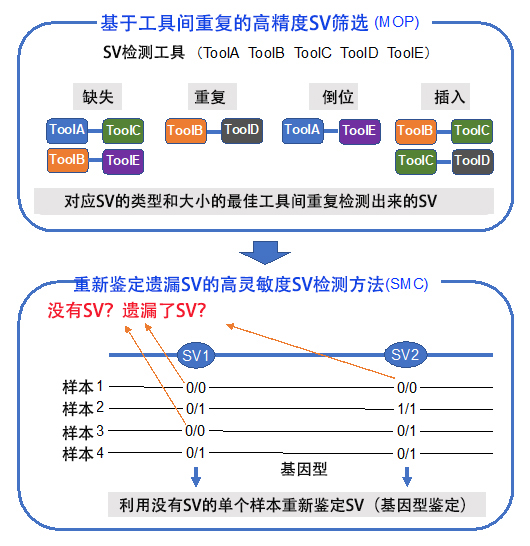
图1 本次开发的“MOPline”算法(供图:理化学研究所)
基因组的SV是超过50个碱基对(bp)的缺失、插入、重复、倒位变异的统称,有别于小于50bp的缺失、插入对应的插入缺失(indel),以及单个碱基置换的单碱基变异(SNV)。大量研究表明,导致个体基因组之间存在巨大差异的SV是包括发育障碍和智力障碍在内的各种人类疾病和性状的遗传因素。
另一方面,由于SV结构复杂和尺寸较大,其检测比SNV更加困难。基因组多态性通常通过将100~150bp的短序列(读取)数据与标准人类基因组序列(参考序列)进行对齐来检测。虽然SNV和插入缺失可纳入读取长度内,但尺寸较大的SV无法纳入读取盒内,所以必须使用跨SV对齐读取的间接证据检测,从而导致检测精度和灵敏度降低。
尽管目前已经开发出多种SV检测工具,但一直存在检测结果通用性较低的问题,还没有能够通过单一工具实现高精度和高灵敏度SV检测的工具。
研究团队发现,能够进行高精度检测的重叠SV(现有SV检测工具间共同检测出的SV)并没有表现出高精度,所以研究团队调查了现有工具的最佳组合。然后,开发了使用现有的4~9个工具按SV类型和大小决定最佳工具组合的算法,并将其命名为MOP(Merging Overlap calls from selected Pairs of algorithms)。
若使用MOP,虽然可以鉴定具有高精度的SV,但会漏掉一部分SV。因此,通过扫描用MOP未能检测出SV的基因组区域,确认了SV的存在。在这一存在确认中,使用了一种利用读取对齐信息的独特基因型鉴定方法,这种重新鉴定SV的方法被命名为SMC(Supplementing Missing Calls)。
最终,研究团队成功开发出结合MOP、SMC、过滤和标注功能的SV检测方法——MOPline。
使用全基因组测序数据验证MOPline的SV检测精度和灵敏度时,显示其结果超过了现有工具的精度和灵敏度。此外,使用从公共数据库(千人基因组计划)获得的100个全基因组测序数据,将多个工具组合与检测SV的现有管道进行了比较。结果显示,MOPline的SV检测精度与现有工具相当,但检测到的真阳性SV(尤其是插入)的数量(检测灵敏度)超过了现有工具。
其次,使用MOPline从3258人的BBJ全基因组测序数据中检测SV后发现,检测到出大约13万4000(个人平均约1万6000)的SV,这一数字比迄今为止的大规模SV研究项目中检测到的个人平均SV数量高出1.7~3.3倍。
该BBJ全基因组测序数据来自至少患有癌症和痴呆症等中的一种疾病的患者。因此,对与疾病相关的已知基因的蛋白质编码区域重叠的SV进行调查后发现,几种罕见的SV与疾病样本中特异的已知疾病风险基因(大肠癌、乳腺癌等)的蛋白质编码区域重叠。
以MOPline检测到的BBJ-SV为参照面板,对18万人的SNP阵列数据(SNP基因型数据)进行填补,推断出约18万人的SV。使用推断的SV和约18万人的医疗信息,对42种疾病和60种定量性状进行了全基因组关联分析(GWAS)后发现,针对包括癌症等疾病在内的32种性状,发现了41个与SNP具有同等或更强相关性的SV。在相关的SV中,8个SV与相关基因的编码区重叠,其中5个是之前未报道与该性状相关的新鉴定的SV。
MOPline是一种展示了前所未有的SV检测精度和灵敏度的工具,不仅可以鉴定导致单基因疾病的罕见SV,还可以通过对SV进行填补,鉴定与数量性状相关的SV。
原文:《科学新闻》
翻译:JST客观日本编辑部
【论文信息】
杂志:Cell Genomics
论文:Detection of trait-associated structural variations using short read sequencing
DOI:10.1016/j.xgen.2023.100328